Los mecanismos pueden ser intrínsecos o adaptativos. Los primeros pueden capacitar a la bacteria para que produzca enzimas que destruyan al fármaco antibacteriano, expresar sistemas efflux de excreción que eviten que el fármaco alcance su blanco intracelular, modificar el sitio blanco del antimicrobiano o generar una vía metabólica alterna que evite la acción del fármaco. Entre los mecanismos adaptativos, encontramos las adaptaciones fenotípicas, sea por el estado metabólico de la bacteria, o por ser secundaria a su capacidad de producir biopelículas.
Las bacterias pueden ser resistentes intrínsecamente a uno o más agentes antimicrobianos, pueden adquirir la resistencia por mutaciones de novo o por genes de resistencia de otros organismos, así como por adaptaciones metabólicas al fármaco.
Resistencia intrínseca
La adquisición de material genético por las bacterias susceptibles a antimicrobianos de bacterias con resistencia ocurre a través de conjugación, transformación o transducción con transposones que a menudo facilitan la incorporación de genes de resistencia múltiple al genoma o plásmido. El uso de agentes antimicrobianos también crea una presión selectiva para el surgimiento de cepas resistentes S. aureus es un ejemplo claro de esta situación. En la era previa a los antibióticos, la mortalidad de pacientes era del 80% al 70%, y para la década de 1940, con la llegada de la penicilina, el pronóstico de los pacientes con infecciones estafilocócicas era muy alentador.
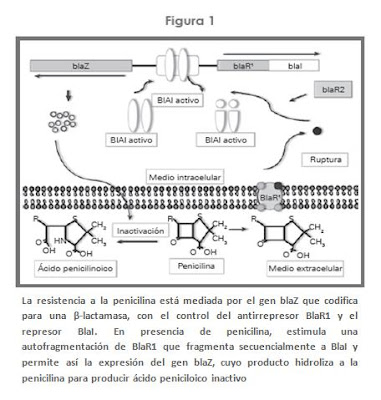
La resistencia a la penicilina está mediada por el gen blaZ, cuyo producto es la β-lactamasa, que hidroliza el anillo β-lactámico de la penicilina y lo inactiva (Figura 1)
En este contexto surge la meticilina, la primera penicilina semisintética resistente a la β-lactamasa, pero rápidamente se reportaron cepas resistentes a la meticilina (MRSA). El gen mecA es el responsable de la resistencia a la meticilina, que forma parte de un elemento genético móvil encontrado en todas las cepas resistentes a la meticilina. Con este mismo comportamiento, se generaron cepas resistentes a las quinolonas, aminoglucósidos y cefalosporinas, con la identificación de los genes responsables en cada caso.Con la llegada de la vancomicina, las cepas MRSA se trataban con éxito y el uso de este fármaco se incrementó para tratar infecciones estafilocócicas resistentes a la meticilina
Los microorganismos P. aeruginosa, M. tuberculosis, S. pneumoniae, S. epidermidis, E. coli y K. neumoniae de igual manera, a lo largo de la historia, han mostrado adaptaciones a muchos de los antibióticos actualmente disponibles y se han identificado los genes responsables en cada adaptación. En estos casos, podemos mencionar al grupo de genes Bla, que produce las resistencias a beta-lactámicos de espectro ampliado y cefalosporinas, los erm A, B y C para macrólidos, mecA meticilina, etc. Todos estos microorganismos causan más del 60% de las infecciones hospitalarias y un porcentaje considerable de las comunitarias.
Multirresistencia (MR) mediada por bombas de excreción efflux
Otro mecanismo de resistencia importante es evitar que el antibiótico se incorpore a la célula bacteriana y lo exporte activamente al medio extracelular, sin permitir el alcance de la molécula diana por el fármaco (bombeo de excreción efflux [BE]).
Los genes y proteínas de las BE están presentes en todos los organismos. En las bacterias, los genes que codifican las bombas efflux se localizan en el cromosoma o los plásmidos.
Existen cinco superfamilias de proteínas de BE:
- Familia de casete de unión al ATP (ABC, por sus siglas en inglés, ATP binding cassette);
- Superfamilia del facilitador mayor (MFS, major facilitator superfamily);
- Familia de extrusión de multifármacos y tóxicos (MATE, multidrug and toxic-compound extrusion);
- Familia de resistencia pequeña a multifármacos (SMR, small multidrug resistance)
- Familia de resistencia a división por nodulación (RND, resistance nodulation division) (Figuras 2 y 3).
Un solo organismo puede expresar más de una familia de BE. En el caso de P. aeruginosa y E. coli, pueden expresar más de un tipo de BE de la familia RND, las cuales se expresan por bacterias Gram negativas y se relacionan con multirresistencias clínicamente significativas
En bacterias Gram negativas, TolC puede funcionar como el canal proteico para las diferentes BE miembros de la familia RND, también pueden interactuar con transportadores MFS y la superfamilia ABC.13
Algunos agentes antibacterianos no son útiles para el tratamiento de infecciones por algunas bacterias Gram negativas, pues éstas tienen resistencia intrínseca a estos agentes. Tal resistencia se atribuyó al principio a la deficiente permeabilidad de la membrana bacteriana a la droga, lo cual ya se ha reportado e indica que la resistencia intrínseca de P. aeruginosa a varios antibióticos se debe a BE.12
La BE es el mecanismo de sospecha de la resistencia antimicrobiana cuando se incrementa la concentración mínima inhibitoria (CMI) de tres o más antibióticos para una bacteria en particular, en comparación con la CMI de estos antibióticos frente a la cepa nativa. La CMI de estos agentes antimicrobianos para BE mutante puede ser de dos a ocho veces más alta que la CMI de estos agentes para la cepa susceptible. Cuando el CMI se incrementa más de 100 veces, por lo general se asocia con cualquier expresión de enzimas que inactive agentes antimicrobianos
Resistencia adaptativa a antibióticos
Indiferencia al fármaco
Las bacterias que no se están dividiendo no son eliminadas por los fármacos. Este fenómeno, que Walsh McDermott denominó “indiferencia al fármaco”, no se limita a la familia de antibióticos β-lactámicos.38-40 Las bacterias que no se dividen, y/o no tienen suficientes nutrientes para un metabolismo activo, son parcial o completamente resistentes a antibióticos bactericidas.
Este efecto resistente no-replicante, en subpoblaciones genéticamente susceptibles de bacterias a tratamiento con antibióticos, es particularmente bien conocido para el tratamiento de infecciones con Mycobacterium tuberculosis
Persistencia
Es conocido que los antibióticos bactericidas no eliminan todas las bacterias aun en crecimiento activo.45 Conforme pasa el tiempo, el rango de eliminación declina en fracción sustancial de la población bacteriana que sobrevive al encuentro con ese fármaco. Este fenómeno se denomina persistencia bacteriana, resistencia adaptativa o tolerancia fenotípica.
La razón por la cual la subpoblación persistente puede sobrevivir a la exposición de antibióticos parece la misma condición que la población a la cual le es indiferente el fármaco: las células bacterianas no estaban en replicación al momento de su exposición al fármaco.
Se han propuesto varios mecanismos para explicar por qué deja de replicarse un subconjunto de bacterias (SOS systems) y así adopta el fenotipo de resistencia.48 Otro mecanismo sería que, durante el curso de crecimiento, las poblaciones bacterianas incluyesen células en proceso de reparación de su ADN y no se estuviesen dividiendo, lo que generaría un fenotipo resistente
Biopelículas
Aunque los genes de resistencia y sus productos son los principales mecanismos de resistencia a antibióticos existen otros menos explorados, como el relacionado con la producción de biopelículas. Las biopelículas son agregados adherentes que se forman en superficies bióticas o abióticas.50 Se ha demostrado que las cepas que producen biopelículas son significativamente más resistentes a antibióticos y agresores antimicrobianos, incluso con respuesta del hospedero
Esta forma de crecimiento bacteriano contiene células genéticamente sensibles, refractarias a muchos antibióticos, pero no a todos.53,54 Se han propuesto tres mecanismos por los cuales las biopelículas contribuyen a la resistencia.
Primero, se postulado que las células bacterianas encajadas en matrices de polisacáridos que constituyen la biopelícula son menos accesibles a la difusión del antibiótico.55 La segunda razón es que se trata de una forma de indiferencia el fármaco a causa de los nutrientes y otros limitantes, pues muchas células bacterianas dentro de la biopelícula no se replican ni metabolizan lo suficiente para que el antibiótico funcione de manera eficaz.56,57 La tercera hipótesis, y actualmente la más apoyada, es que las bacterias dentro de las biopelículas se diferencian a estados refractarios a los antibióticos; es decir, una combinación de indiferencia y persistencia, factores ya comentados.58
Esto debe considerarse en el tratamiento. Por ejemplo, P. aeruginosa produce biopelículas donde los antibióticos, como las fluoroquinolonas, penetran más fácil en comparación con los aminoglucósidos, pues éstos se unen a polímeros como el alginato,59 además de considerar interacciones muy importantes, como la de los aminoglucósidos como inductores de la formación de biopelículas en cepas de P. aeruginosa y E. coli, lo que exacerba la resistencia.